Les grands projets de séquençage sont fondamentaux pour mieux comprendre le vivant dans différents domaines (santé, agronomie, écologie). Les avancées technologiques ont permis d’atteindre une taille considérable de données brutes de séquençage (lectures de séquences). Le service européen « European Nucleotide Archive » contient actuellement près de 50 Petaoctets de données brutes publiques.
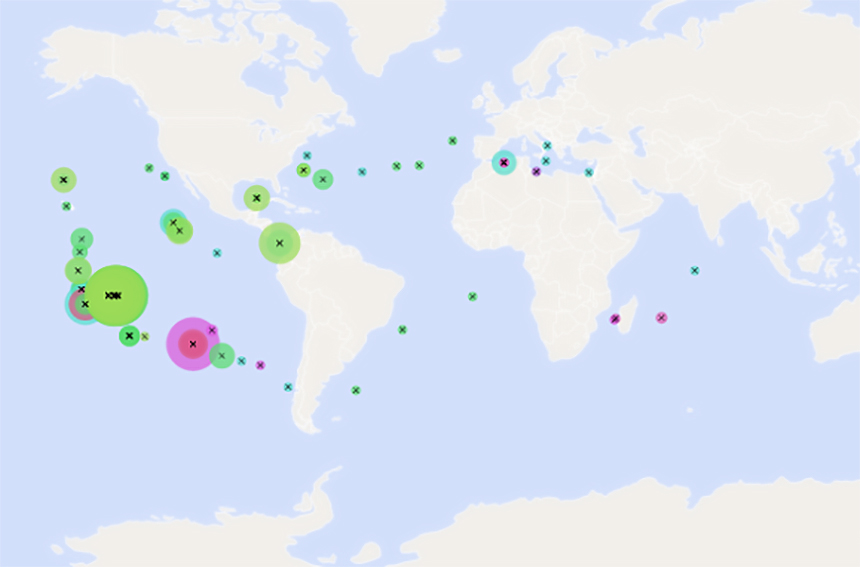
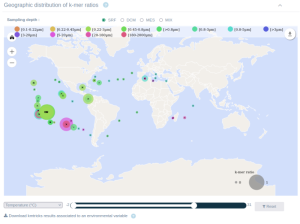
Une équipe de chercheurs du CNRS Terre & Univers, en collaboration avec plusieurs laboratoires de recherche, a utilisé des k-mers (mots de taille k) pour créer une notion de mot dans les données brutes de séquençage. Cette solution d’indexation a ainsi permis d’interroger plusieurs dizaines de téraoctets de données de séquence issues du projet Tara Oceans. Le serveur web public Ocean Read Atlas (ORA), développé pour ce propos, permet d’interroger directement plusieurs jeux de données du consortium Tara Oceans prélevés sur tous les océans du globe. À l’aide d’une ou plusieurs séquences, il est possible d’identifier la présence de séquences similaires sous forme de carte et de graphique interactifs (voir figure 1) dans les stations de prélèvement en fonction de leurs propriétés environnementales (température, salinité, oxygène, etc.).
Auparavant, une requête de 1000 séquences nécessitait plusieurs semaines de calculs sur des supercalculateurs, la réponse est désormais instantanée avec ORA sans compresser la structure des données. Cette étude ouvre ainsi de nouvelles perspectives dans le domaine de la génomique et l’écologie numérique grâce à l’accès et l’exploitation des dizaines de téraoctets de données génétiques. Ce modèle d’indexation de données de séquençage sera utilisé pour les campagnes océanographiques BioSWOT-Med et APERO.
Magali Lescot, Ingénieure de recherche MIO
Cyrille Blanpain, Responsable du Service Informatique de l’OSU Pythéas